Ultragevoelige biosensor voor de detectie van Vibrio cholerae DNA met polystyreen-co-acrylzuur composiet nanosferen
Abstract
Een ultragevoelige elektrochemische biosensor voor de bepaling van pathogene Vibrio cholerae (V . cholerae ) DNA is ontwikkeld op basis van polystyreen-co-acrylzuur (PSA) latex nanosferen-gouden nanodeeltjes composiet (PSA-AuNP's) DNA-dragermatrix. Differentiële pulsvoltammetrie (DPV) met behulp van een elektroactief antraquninon-oligonucleotidelabel werd gebruikt voor het meten van de biosensorrespons. Het laden van gouden nanodeeltjes (AuNP's) op de DNA-latex-deeltjeselektrode heeft de faraday-stroom van DNA-hybridisatie aanzienlijk versterkt. Samen met het gebruik van een gerapporteerde sonde heeft de biosensor een hoge gevoeligheid aangetoond. De DNA-biosensor leverde een reproduceerbaar en breed lineair responsbereik op om DNA te targeten van 1,0 × 10 −21 tot 1.0 × 10 −8 M (relatieve standaarddeviatie, RSD = 4,5%, n = 5) met een detectielimiet (LOD) van 1,0 × 10 −21 M (R 2 = 0.99). De biosensor behaalde bevredigende herstelwaarden tussen 91 en 109% (n = 3) voor de detectie van V . cholerae DNA in verrijkte monsters en kan worden hergebruikt voor zes opeenvolgende DNA-assays met een herhaalbaarheid RSD-waarde van 5% (n = 5). De reactie van de elektrochemische biosensor was stabiel en handhaafbaar op 95% van de oorspronkelijke reactie tot een bewaarperiode van 58 dagen.
Achtergrond
Vibrio cholerae , een door voedsel overgedragen ziekteverwekker, kan epidemieën van cholera bij mensen veroorzaken door de toestand van acute waterige diarree. Cholera-uitbraak is nog steeds een ernstig probleem in sommige delen van de wereld, b.v. Azië en Afrika, en leidt tot een lage sociaaleconomische status [1,2,3,4]. Deze darmpathogeen is een belangrijke oorzaak van morbiditeit en mortaliteit, vooral in ontwikkelingslanden [5]. De epidemie en pandemie van cholera in verschillende regio's wordt voornamelijk veroorzaakt door V. cholerae serogroepen O1 en O139 [1, 2, 6]. V. cholerae serogroep O1 heeft twee belangrijke serotypen, namelijk Inaba en Ogawa, die elkaar kunnen afwisselen tussen cholera-epidemieën. Het derde serotype, Hikojima, bestaat ook, maar is zeldzaam en onstabiel. De genen die verantwoordelijk zijn voor de biosynthese van O1-antigeen zijn aangeduid als rfb. De mutatie die de serotypen Inaba en Ogawa definieert, is een enkele deletiemutatie in het rfbT-gen [7]. Er is echter ook gemeld dat incidentele door voedsel overgedragen uitbraken bij mensen met ernstige diarree worden veroorzaakt door niet-O1/niet-O139 V. cholerae door de inname van onvoldoende verhitte zeevruchten [8] of blootstelling aan een verontreinigd aquatisch milieu [9]. De eerste epidemie van V. cholerae O139 kwam voor in 1992 in Bangladesh en India en verspreidde zich daarna snel naar andere landen in Zuidoost-Azië [10]. In 2005 werden wereldwijd in totaal 131.943 choleragevallen en 2272 sterfgevallen gemeld aan de Wereldgezondheidsorganisatie (WHO) [11].
De zoektocht naar een effectieve methode voor monitoring of diagnose van toxigene V. cholerae bacteriën zijn absoluut noodzakelijk om de cholera-epidemie onder controle te krijgen. Traditionele identificatie van V. cholerae wordt vaak bereikt door de isolatie en screening van de bacteriën, waarbij het gaat om voorverrijking in alkalisch peptonwater (APW) gevolgd door isolatie van V. cholerae op het thiosulfaat citraat galzout sucrose agar (TCBS) kweekmedium en identificatie door middel van agglutinatietest op objectglaasjes met specifieke antisera [12]. Deze techniek is echter zeer tijdrovend en arbeidsintensief, en het resultaat dat enkele dagen later wordt verkregen, zou een vertraging in de klinische diagnose en de behandeling van de patiënt hebben betekend. Moleculaire methode met PCR-amplificatie voor de detectie van V. cholerae [13] heeft de diagnosetijd verkort [14], maar de PCR-methode vereist bekwame professionele en dure infrastructuur die moeilijk uit te voeren is in landen met weinig middelen. Er is melding gemaakt van snelle diagnostische tests op basis van het principe van immunochromatografie voor discrete of gelijktijdige detectie van V. cholerae serogroepen O1 en O139. Enkele andere op immunoassay gebaseerde technieken die worden gebruikt voor de detectie van V. cholerae zijn zoals enzyme-linked immunosorbent assay (ELISA), coagglutinatie, immunofluorescentie en kwartskristalmicrobalans (QCM). De meeste van deze technieken vereisen echter geavanceerde instrumenten, een lange testtijd en hooggekwalificeerd personeel met gedetailleerde technische kennis [15,16,17,18,19,20].
Elektrochemische methoden hebben veel aandacht getrokken bij de detectie van nucleïnezuur vanwege hun hoge gevoeligheid, specificiteit, eenvoud en economisch protocol, evenals snelle detectie en compatibel met microfabricagetechnologie [21, 22]. Bovendien kan een elektrochemische methode die is gekoppeld aan miniaturisatietechnologieën worden gebruikt voor in situ gedecentraliseerde analyse, bijvoorbeeld het op microfluïdische chip gebaseerde DNA-biosensorapparaat, wat erg handig is voor praktische instellingen [23]. Er is een breed scala aan elektroden die worden gebruikt bij de elektrochemische metingen, zoals een glasachtige koolstofelektrode (GCE), een koolstofpasta-elektrode (CPE), een goudelektrode en een platina-elektrode. Onlangs zijn studies geconcentreerd op het gebruik van gezeefdrukte elektroden (SPE's) vanwege enkele van hun unieke eigenschappen, zoals het bieden van een lage achtergrondstroom en een breed potentiaalvenster, kosteneffectief omdat de koolstofinkt goedkoop is en in massa kan worden geproduceerd .
Er zijn enkele elektrochemische methoden gerapporteerd voor de detectie van V. cholerae bestond uit een reeks complexe stappen. Een elektrochemische V. cholerae genosensor gerapporteerd door Liew et al. [24] gebruikte elektrochemische adsorptiemethode om de DNA-sonde op de koolstof-SPE te immobiliseren. De gelyofiliseerde AuNPs-gemodificeerde meerlaagse PSA-deeltjes met polyelektrolyten vormden bioconjugaat met avidine om te functioneren als het reporterlabel in de sandwich-DNA-hybridisatietest. De toevoeging van sorbitolstabilisator was echter nodig om de PSA-AuNPs-avidine-bioconjugaten te behouden om de operationele periode van de DNA-biosensor tot 30 dagen te verlengen. Enzymatische elektrochemische V. cholerae DNA-biosensor is onlangs bedacht door Yu et al. [25], waarbij de gethioleerde anti-fluoresceïne-geconjugeerde alkalische fosfatase (anti-FCAP)-gelabelde DNA-probe werd gebonden aan de gouden SPE door middel van goud-thiol-chemie. Het doelwit-DNA werd gelabeld met een universele fluoresceïne om DNA-hybridisatieherkenning mogelijk te maken via enzymatische omzetting van α-naftylfosfaat in elektroactief α-naftol. Desalniettemin vereiste dit detectieschema een lange testtijd van ongeveer 95 minuten voor DNA-hybridisatie, labeling van de DNA-hybriden met functioneel enzym gevolgd door incubatie van de elektrode in elektro-inactief α-naftylfosfaatsubstraat voordat een amperometrische meting kan worden uitgevoerd. Een andere enzymatische elektrochemische DNA-biosensor op basis van met avidine gecoate koolstof SPE geconjugeerd met gebiotinyleerde DNA-sonde werd ontwikkeld door Low en teamleden [26]. Een met digoxigenine (DIG) gemerkte reporterprobe werd ook gebruikt in deze dubbele hybridisatiestrategie die de cDNA-sequentie flankeerde. Mierikswortelperoxidase-gekoppelde anti-DIG (anti-DIG-HRP) werd gebruikt als het elektrochemische label, dat tegelijkertijd de oxidatie van 3,3′,5,5′-tetramethylbenzidine (TMB) kon katalyseren met de reductie van H 2 O2 om een elektronenoverdracht naar het elektrodeoppervlak op te leveren voor elektrochemische transductie van DNA-hybridisatiegebeurtenis. Een eenvoudig DNA-biosensorontwerp op basis van gethioleerde DNA-sonde geïmmobiliseerde goudgecoate glazen elektrode werd beschreven door Patel et al. [22] voor snelle detectie van V. cholerae en methyleenblauw werd gebruikt als de DNA-hybridisatie-indicator. Het lineaire detectiebereik van het systeem is echter beperkt tot μM-niveaus, wat de toepassing ervan in klinische monsters beperkte.
Latex-goud nanodeeltjes zijn eerder gebruikt als het DNA-hybridisatielabel via avidine/biotine-binding aan de DNA-probe bij de detectie van V. cholerae [24], vispathogeen Aphanomyces invadans [27], E. coli [28], en niet-specifieke DNA-hybridisatie [29], waarbij de latexbolletjes werden gecoat met een meerlaagse polyelektrolyt voordat de negatief geladen colloïden van gouden nanodeeltjes er elektrostatisch aan werden bevestigd. Kawde en Wang [29] bevestigden de PSA-latexdeeltjes aan een DNA-reportersonde om te worden gebruikt als DNA-hybridisatielabel door het laden van met streptavidine gecoate latexdeeltjes met biotine-gecoate AuNP's. Kuan et al. [24, 27] en Liew et al. [24, 27] rapporteerde ook dezelfde avidine-biotine-bindingsmethode met behulp van goud-PSA-DNA-probeconjugaten. Pinijsuwan et al. [28] rapporteerde het gebruik van een elektrostatische methode voor het laden van PSA-deeltjes gehecht aan DNA-reportersonde en de met polyelektrolyt goud beklede PSA-deeltjes werden gebruikt als een label voor hybridisatie die de DPV-stroomrespons versterkte.
In deze studie rapporteren we een andere DNA-immobilisatiebenadering met behulp van de latex-gouden nanodeeltjes als het DNA-probe-immobilisatiesubstraat om een zeer gevoelig detectiesysteem voor V te ontwikkelen. cholerae DNA. Immobilisatie van DNA werd uitgevoerd met een zeer eenvoudige en snelle procedure met behulp van 1-ethyl-3-(3-dimethylaminopropyl) carbodiimide hydrochloride/N-hydroxysuccinimide (EDC/NHS) chemie als koppelingsreagens voor het verbeteren van de immobilisatie-efficiëntie [30] op de gecarboxyleerde latex deeltjes oppervlak. De detectie van DNA-hybridisatie was gebaseerd op de sandwich-type test, die een hybridisatiereactie tussen geïmmobiliseerde DNA-probe en doelsequentie inhield, gevolgd door een signaal/reporter-probe. Antrachinon-2-sulfonzuur-monohydraat-natriumzout (AQMS) werd gebruikt als een elektrochemisch label om de hybridisatie te volgen. De voorgestelde latexdeeltjes van submicron grootte verbeterden de bindingscapaciteit van de DNA-sonde en de gevoeligheid van de DNA-biosensor werd verbeterd door de opname van de zeer geleidende gouden nanodeeltjes (AuNP's). De DNA-biosensor vertoonde een uitzonderlijke gevoeligheid voor de detectie van V. cholerae cDNA en extreem lage detectielimiet op zeptomolaire niveaus vergeleken met tot nu toe gerapporteerde avidine-biotine-technologie [24, 26].
Methoden
Chemische stoffen en reagentia
Styreen (St) en acrylzuur (AA) werden gekocht bij Fluka. HAuCl4 ·3H2 O, trinatriumcitraat-dehydraat, natriumdodecylsulfaat (SDS), 1-ethyl-3-(3-dimethylaminopropyl)carbodiimide-hydrochloride (EDC) en N-hydroxysuccinimide (NHS) werden verkregen van Sigma-Aldrich. Ammoniumpersulfaat (APS), waterstofbromide en broom werden respectievelijk geleverd door Riedel-De Haën, Ajax Finechem en Panreac. Alle chemische oplossingen werden bereid met gedeïoniseerd water. Zowel 30-base doelwit als mismatch synthetische oligonucleotiden werden verkregen van Bio Service Unit (NSTDA). Niet-complementair DNA (ncDNA) en signaalprobe waren van Sigma en de 5'-amino-gemodificeerde invangprobe was van Bioneer. De vangprobe werd bereid in 0,05 M kaliumfosfaatbuffer (pH 7), terwijl doel-DNA, niet-overeenkomende doel-, reporterprobe en niet-complementaire DNA-oplossingen werden bereid in natriumfosfaatbuffer (0,05 M, pH 7). De oligonucleotidesequenties die in de huidige studie zijn gebruikt, worden weergegeven in tabel 1.
Apparaat
De elektroanalytische meting werd uitgevoerd met een potentiostaat/galvanostaat (Autolab PGSTAT12, Metrohm) uitgerust met GPES (4.0.007) software. De voltammetrische experimenten werden uitgevoerd met een conventioneel systeem met drie elektroden, bestaande uit een met koolstofpasta gezeefdrukte werkelektrode (SPE) (Quasense, Bangkok, Thailand), Ag/AgCl-referentie-elektrode (3 M KCl) en platinastaaf ( 2 mm diameter) tegenelektrode. Differentiële pulsvoltammetrie (DPV) techniek werd gebruikt voor alle elektrochemische onderzoeken bij 0,02 V stappotentiaal en 0,5 V/s scansnelheid van 0,0 tot +1,0 V in 4,5 ml meetbuffer (0,05 M kaliumfosfaatbuffer) bij pH 7 en omgevingstemperatuur. Alle potentialen die in dit onderzoek werden gemeten, werden verwezen naar Ag/AgCl-elektroden en homogene oplossingen werden bereid met behulp van een ultrasoonapparaatbad (Elma S30H). Een scanning elektronenmicroscoop (SEM, LEO 1450VP) werd gebruikt om de grootte en verdeling van de latexbolletjes te bepalen.
Methoden
Synthese van colloïdale gouden nanodeeltjes
Colloïdale AuNP's werden bereid door natriumcitraatreductie volgens de Turkevich-methode [31]. In het kort, ongeveer 10 ml van 5 mM HAuCl4 ·3H2 O werd opgelost in 180 ml gedeïoniseerd water en onder continu roeren gekookt op een gecombineerde hete plaat-magnetische roerinrichting. Tien milliliter van 0,5% (w /v ) trinatriumcitraat werd vervolgens aan de kokende oplossing toegevoegd en de kleur van de oplossing werd geleidelijk veranderd van lichtrood naar robijnrood.
Bereiding van latexdeeltjes
Latexdeeltjes werden bereid door een zeepvrije emulsiecopolymerisatiereactie zoals beschreven door Polpanich et al. [23] met enkele aanpassingen. In het kort, ongeveer 190 g gedeïoniseerd water werd gespoeld met stikstofgas in een driehalskolf ondergedompeld in een waterbad gedurende -1 uur onder roeren bij 350 rpm. Twintig gram St en 0,5 g AA werden vervolgens toegevoegd en de temperatuur werd op 70°C gehouden. Daarna werd ongeveer 0,2 g APS toegevoegd aan 10 ml gedeïoniseerd water, gevolgd door gieten in de formulering in de driehalskolf om de radicaalpolymerisatiereactie te initiëren, en het polymerisatieproces werd gedurende 8 uur voortgezet. De as-gesynthetiseerde carboxyllatexbolletjes werden geoogst door tweemaal 20 min centrifugeren met gedeïoniseerd water bij 13.000 rpm [23, 27, 28] en opnieuw gesuspendeerd in gedeïoniseerd water bij kamertemperatuur (25°C) tot gebruik. De morfologie en gemiddelde grootte van de PSA-latexdeeltjes werden bepaald door middel van scanning elektronenmicroscopie (SEM).
Wijziging van SPE-oppervlak en DNA-sonde-immobilisatie
Voorafgaand aan oppervlaktemodificatie werd de koolstof-SPE grondig gespoeld met gedeïoniseerd water en vervolgens gedruppeld met de PSA-suspensie van 3 mg / ml en aan de lucht gedroogd bij omgevingscondities gevolgd door drop-casting met 5 mg / ml colloïdale AuNP's . De elektrochemische kenmerken van koolstof SPE voor en na modificatie met latexdeeltjes en AuNP's werden onderzocht met de CV-methode. De latex-AuNPs-gemodificeerde koolstof SPE (PSA-AuNPs-SPE) werd vervolgens gespoeld met gedeïoniseerd water en ondergedompeld in 0,1 M kaliumfosfaatbuffer (pH 5) die carbodiimide-verknopingsreagentia bevat, dwz 0,002 M EDC en 0,005 M NHS gedurende 2 uur [32] alvorens een nacht te weken in 0,05 M kaliumfosfaatbuffer (pH 7) die 5 μM vangsonde bevat. Daarna werd de DNA-gemodificeerde PSA-AuNPs-SPE (DNA-PSA-AuNPs-SPE) grondig gewassen met kaliumfosfaatbuffer (0,05 M, pH 7) om de fysiek geadsorbeerde sonde te verwijderen. De DNA-elektrode werd gedurende 1 uur ondergedompeld in 0,05 M natriumfosfaatbuffer bij pH 7 met lineair doel-DNA (1 M) en AQMS DNA-hybridisatielabel (5 mM) voor gedeeltelijke hybridisatie en later gedompeld in 0,05 M natriumfosfaatbuffer (pH 7) geconditioneerd met 1 M reporterprobe en 5 mM AQMS gedurende nog eens 1 uur voor een volledig DNA-hybridisatieproces. Ten slotte werd de DNA-elektrode gespoeld met kaliumfosfaatbuffer (0,05 M, pH 7) voor de verwijdering van niet-gehybridiseerde oligonucleotidesequenties. De elektrochemische respons van elke verlengde stof die op de SPE was bevestigd, werd onderzocht met de DPV-methode. Figuur 1 geeft de stapsgewijze procedure weer voor de ontwikkeling van V. cholerae DNA-biosensor op basis van colloïdale PSA-AuNP's solide ondersteuning.
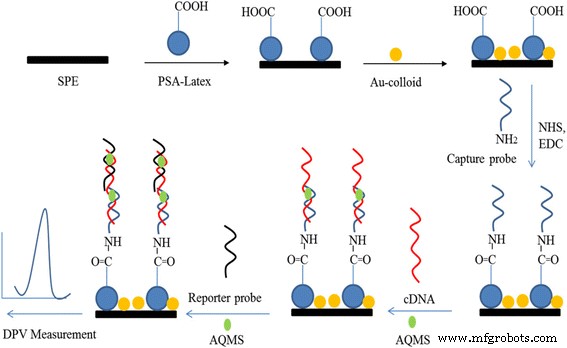
Schematische weergave van stapsgewijze fabricageprocedure van DNA-biosensor
Optimalisatie van hybridisatie van synthetische oligonucleotiden
Voorafgaand aan de bepaling van het dynamisch lineair bereik van het DNA is het effect onderzocht van verschillende parameters zoals DNA-probe en AQMS-concentraties, pH, ionsterkte en buffercapaciteit op de hybridisatierespons van de geïmmobiliseerde DNA-probe met signaalprobe en doel-DNA biosensor. Bovendien werden de immobilisatie- en DNA-hybridisatieduur van de capture-probe, evenals de levensduur en regeneratie van de biosensor ook beoordeeld vóór de ontwikkelde V. cholerae DNA-biosensor was klaar voor toepassing in het spike-and-recovery-experiment. Capture-probe en AQMS-ladingen werden geoptimaliseerd door hun concentraties te variëren van respectievelijk 1 tot 6 M en 0, 1-5 mM, terwijl de concentratie van doel-DNA en reportersonde op 5 μM werd gehouden in 0, 05 M natriumfosfaatbuffer (pH 7, 0). Onderzoeken naar het pH-effect en de bufferconcentratie werden uitgevoerd door de pH en de concentratie van natriumfosfaatbuffer te veranderen van respectievelijk pH 5,5 tot 8,0 en 0,001 tot 1.000 M. De aanwezigheid van verschillende kationen in de DNA-hybridisatiereactie van de elektrochemische DNA-biosensor werd uitgevoerd door Na + toe te voegen. , K + , Ca 2+ , en Fe 3+ ionen bij 1,0 M in de DNA-hybridisatiebuffer met 1 mM AQMS en 5 μM cDNA en detectieprobe met pH 7,0. Het effect van ionensterkte werd onderzocht door de NaCl-concentratie te variëren over het bereik van 0,1-3,0 M bij pH 7,0. Dynamisch bereik van de DNA-biosensor werd vervolgens bepaald in 1,0 × 10 −21 tot 1.0 × 10 −8 M V. cholerae cDNA met behulp van een constante signaalprobe-concentratie bij 5 M en pH 7,0. De immobilisatieduur van de DNA-sonde werd bepaald door de DNA-elektrode tussen 1 en 13 uur in 5 μM vangsonde-oplossing (pH 7, 0) te weken en de DPV-respons werd elke 1-2 uur genomen. Ondertussen werd de DNA-hybridisatietijd bepaald door de DNA-hybridisatiereactie tussen 15 en 180 minuten te laten plaatsvinden, en de DNA-biosensorrespons werd elke 15-30 minuten geregistreerd. De houdbaarheid van de DNA-biosensor werd bepaald door periodiek de DNA-biosensorrespons op de detectie van 5 μM V te meten. cholerae cDNA gedurende 120 dagen. De analyse werd uitgevoerd in vijf herhalingen met gebruikmaking van een nieuwe DNA-elektrode voor elke sandwichhybridisatietest. Regeneratie van de DNA-elektrode werd uitgevoerd met 0,1 M NaOH-oplossing gedurende 4 minuten, en herhybridisatie van de DNA-biosensor (60 minuten) werd bereikt met behulp van herhybridisatie-oplossing die 5 M cDNA en detectieprobe en 1 mM AQMS met een ionsterkte van 2,0 M bevatte in 0,05 M kaliumfosfaatbuffer (pH 7,0). Het regeneratie-experiment werd uitgevoerd in zes herhalingen.
V. cholerae Kwantificering met behulp van op PSA-AuNPs gebaseerde elektrochemische DNA-biosensor
Diverse V. cholerae bacteriestammen namelijk J2126-I, J2126-II, J3324-I, J3324-II, J3330-I, J3330-II, CDHI 5294-II en UVC1324 waaronder Citrobacter freundii (CF-I) en Citrobacter freundii (CF-II) werden verkregen van Microbiology Laboratory, Faculteit Toegepaste Wetenschappen, AIMST University, Kedah. Vervolgens werd genoom-DNA-extractie op deze bacteriën uitgevoerd met behulp van QIAGEN Genomic-tip 500/G volgens het protocol van de fabrikant. Het geëxtraheerde DNA werd vervolgens 100-voudig verdund met behulp van natriumfosfaatbuffer (0,05 M, pH 7,0). Ongeveer 300 ml van het geëxtraheerde DNA dat 2,0 M NaCl en 1 mM AQMS bevatte, werd gedurende 15 minuten gesoniceerd om de DNA-breuken vrij te maken. Vervolgens werd de geïmmobiliseerde DNA-probe gedurende 1 uur geweekt om het DNA-hybridisatieproces te laten plaatsvinden en zorgvuldig gewassen met 0,05 M kaliumfosfaatbuffer (pH 7,0) om het ongebonden DNA te verwijderen. Evaluatie van de DNA-biosensorrespons op basis van DPV-piekstroom werd gemeten en vergeleken met de stroomrespons gegenereerd door de DNA-elektrode zonder reactie met cDNA als controlesignaal. Elk experiment werd in drievoud uitgevoerd onder dezelfde experimentele omstandigheden. Algemeen t test werd gebruikt om een significant verschil te bepalen tussen de reactie van de DNA-biosensor en de reactie van de controle. Het herstel van V. cholerae J3324 en V. cholerae UVC1324 DNA's bij 1.0 × 10 −4 μg μL −1 , 1.0 × 10 −5 μg μL −1 , en 1.0 × 10 −6 μg μL −1 toegevoegd aan de hybridisatiebuffer werd vervolgens uitgevoerd met behulp van de voorgestelde op PSA-AuNPs gebaseerde elektrochemische DNA-biosensor.
Resultaten en discussie
Morfologie van latexdeeltjes en elektrochemisch gedrag van met latex-gouden nanodeeltjes gemodificeerde SPE.
Figuur 2 toont de SEM-microfoto van de gecarboxyleerde latexbolletjes met een gemiddelde deeltjesgrootte van 186,1 ± 4,6 nm. De uniforme grootteverdeling van de PSA-bolletjes maakte homogene immobilisatie van DNA-moleculen op het latexoppervlak mogelijk om de reproduceerbaarheidsrespons van de DNA-biosensor te verbeteren. Een scanning elektronenmicroscoop (SEM, LEO 1450VP) werd gebruikt om de grootte en verdeling van de latexbolletjes te bepalen.

SEM-microfoto van de gesynthetiseerde PSA-latexbolletjes bij 10.000 vergrotingen
De elektrodynamische resultaten van de gemodificeerde SPE zijn weergegeven in tabel 2. De piekpotentiaalscheiding (ΔEp) geeft de elektronenoverdrachtskinetiek van het systeem aan. De PSA-gemodificeerde SPE (PSA-SPE) vertoonde de hoogste ΔEp-waarde vanwege het langzame ladingsoverdrachtproces in de colloïdale copolymeerdeeltjeslaag, waardoor het systeem naar een quasi-reversibele toestand ging. Toen de AuNP's echter op de PSA-SPE werden geladen, impliceert de afname in de ΔEp een verbetering van de elektronenoverdrachtssnelheid op het elektrode-oppervlak.
Figuur 3 toont de differentiële pulsvoltammogrammen van de AQMS-oxidatierespons op de met latex gemodificeerde SPE en de sequentiële hybridisatierespons van de V. cholerae DNA biosensor. Er werd een significant DPV-piekstroomverschil waargenomen tussen elektroden die alleen latex-gemodificeerde microbolletjes bevatten en zonder geïmmobiliseerd capture-DNA (experimenten (a) en (b)) en gemodificeerd met geïmmobiliseerde capture-DNA-sondes in aanwezigheid van cDNA en gerapporteerde sonde (experiment (g )). Dit geeft aan dat de geamineerde DNA-capture-probes met succes werden geïmmobiliseerd op de gecoate gecarboxyleerde PSA-latexbolletjes via EDC/NHS-koppelingsprotocol [33]. Experiment (g) toont ook een veel hogere DPV-respons vergeleken met DNA-elektroden in aanwezigheid van ncDNA en gerapporteerde reporterprobe (experiment (d)), in niet-overeenkomende DNA en gerapporteerde reporterprobe (experiment (e)), en doel-cDNA met geen gerapporteerde sonde (experiment (f)). Dit komt door de volledige hybridisatie van het doelwit-DNA met capture- en reporterprobes door middel van een sandwichhybridisatiereactie op het DNA-biosensoroppervlak, zoals aangetoond in experiment (g). Dit toont ook aan dat het gebruik van de gerapporteerde probe het signaal van DNA-hybridisatie zou kunnen versterken. Desalniettemin is de DPV-stroom die het gevolg is van hybridisatie die is waargenomen in de aanwezigheid van doelwit-DNA zonder opname van een gerapporteerde probe (experiment (f)) nog steeds hoger dan de DPV-stroomsignalen die zijn waargenomen voor niet-gehybridiseerd DNA (experimenten (c), (d) en (e)).
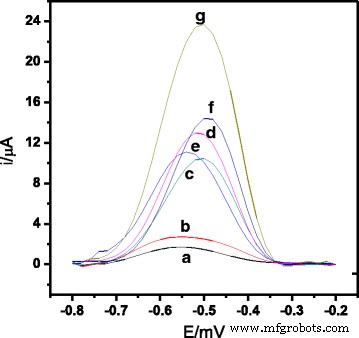
Differentieel pulsvoltammogramsignaal van AQMS van de elektroden (a ) PSA-SPE, (b ) PSA-AuNPs-SPE, (c ) capture probe-PSA-AuNPs-SPE en wanneer in aanwezigheid van (d ) ncDNA en reportersonde, (e ) mismatch DNA en reportersonde, (f ) alleen cDNA, en (g ) cDNA en reportersonde
Effect van het laden van DNA-sondes en AQMS-concentratie
Het effect van de DNA-probeconcentratie op de DNA-hybridisatierespons werd waargenomen via de AQMS elektrochemische oxidatierespons. Figuur 4a laat zien dat de DNA-biosensorrespons geleidelijk toenam met de toename van de hoeveelheid DNA-sonde die op de PSA-AuNPs-SPE was geïmmobiliseerd van 1 tot 4 μM. Dit werd toegeschreven aan de toenemende hoeveelheid elektroactieve AQMS die is geïntercaleerd in het dubbelstrengs DNA (dsDNA) om een elektronenoverdracht door de geïmmobiliseerde DNA-helix mogelijk te maken. Er werd waargenomen dat de DPV-respons van de DNA-biosensor bijna een plateau werd tussen 4 en 6 μM DNA-sonde, wat aangeeft dat een optimale DNA-sondelading op het elektrodeoppervlak werd bereikt [34]. Daarom werd 4 M capture-probe geselecteerd als een optimale DNA-probe-lading in de daaropvolgende experimenten. De concentratie van AQMS-label is ook geoptimaliseerd in de metende elektrolyt tussen 0,1 en 5,0 mM, en de concentratie van AQMS bij 1 mM bleek voldoende te zijn voor een optimale DNA-intercalatiereactie (Fig. 4b).
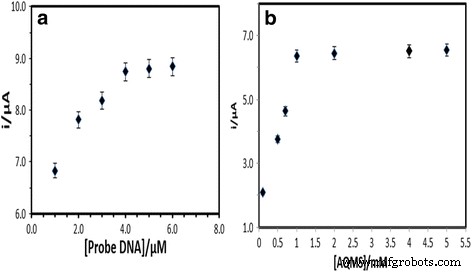
Het effect van de capture-sonde (a ) en AQMS-concentraties (b ) op de DNA-biosensorrespons uitgevoerd met 5 μM cDNA en signaalsonde in 0,05 M natriumfosfaatbuffer (pH 7,0)
Effect van pH, ionsterkte en buffercapaciteit
De snelheid van de DNA-hybridisatiereactie is sterk afhankelijk van de pH van de oplossing. Zoals te zien is in Fig. 5a, verminderde de protonering van de fosfodieter-ruggengraat van DNA onder een meer zure omgeving de oplosbaarheid van het DNA-molecuul, wat uiteindelijk de DNA-hybridisatiereactiesnelheid verminderde. Terwijl het in basisch medium de zwakke waterstofbruggen verbrak die DNA-basenparen bij elkaar hielden. Optimale DNA-hybridisatiereactie was gunstiger in neutrale toestand, waarbij het meer vangprobes bevorderde om te hybridiseren met doel-DNA en vervolgens intercalatie van AQMS-redoxprobes mogelijk maakte om de DNA-hybridisatie-herkenning in de praktijk te brengen. Zo werd 0,05 M natriumfosfaatbuffer bij pH 7,0 gebruikt als het DNA-hybridisatiemedium voor daaropvolgende DNA-biosensoronderzoeken. Positief geladen ionen zoals Ca 2+ , Na + , K + , en Fe 3+ ionen kunnen interageren met de negatief geladen fosfodiesterketen van DNA. Deze ionische reactie zal de lading van het DNA-molecuul neutraliseren, waardoor de sterische afstoting tussen DNA-moleculen wordt verminderd om de DNA-hybridisatiereactie te vergemakkelijken. Figuur 5b geeft het effect weer van enkele kationen op de DNA-hybridisatierespons. Er werd opgemerkt dat de DNA-hybridisatierespons toenam in de aanwezigheid van positief geladen ionen in de orde van Na + > K + > Fe 3+ > Ca 2+ . Beide Ca 2+ en Fe 3+ ionen bleken de DNA-hybridisatierespons aanzienlijk te verminderen vanwege de ionische interacties van Ca 2+ en Fe 3+ ionen met fosfaationen uit de bufferoplossing, wat leidde tot de vorming van onoplosbare fosfaatverbindingen. Dit heeft het ionengehalte van het medium verminderd, waardoor de elektrostatische afstoting tussen DNA-moleculen is toegenomen. De hoogste DNA-hybridisatiestroom werd verkregen in aanwezigheid van Na + ion vanwege zijn kleinere omvang en sterkere affiniteit voor de DNA-suiker-fosfaatruggengraat vergeleken met K + ion om de sterische belemmering en elektrostatische afstoting tussen negatief geladen fosfaatgroepen van DNA's te overwinnen.
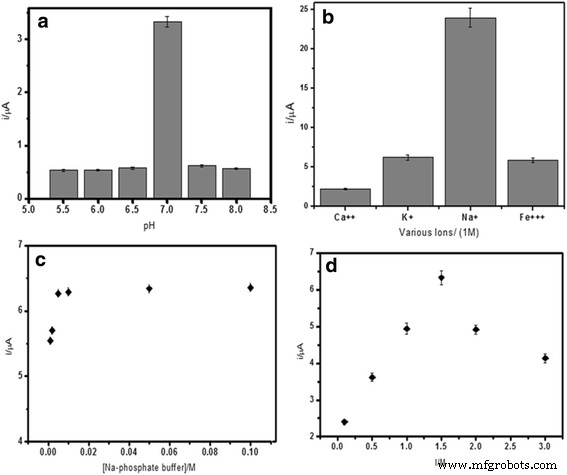
Het effect van pH (a ) verschillende kationen (b ), bufferconcentratie (c ), en ionsterkte (d ) op de DNA-hybridisatiereactie van de elektrochemische V. cholerae DNA biosensor. Hybridisatie werd uitgevoerd met 5 μM cDNA en reporterprobe gevolgd door intercalatie met 1 mM AQMS
Bovendien zou de ionsterkte van de oplossing ook de reactie van de DNA-biosensor beïnvloeden. Figuur 5c, d toont de optimale buffercapaciteit en de ionsterkte werd bereikt met 0,05 M natriumfosfaatbuffer met de pH vastgesteld op respectievelijk pH 7,0 en 2,0 M NaCl. In deze toestand gaf het de meeste voorkeur aan de DNA-hybridisatiereactie; vandaar dat een hoge DPV-respons werd verkregen. Bij optimale buffercapaciteit en ionsterkte van de oplossing nam de elektrostatische afstoting tussen DNA-moleculen af en verbeterde zo de DNA-hybridisatiereactie. Wanneer daarentegen een te laag of te hoog ionengehalte werd gebruikt, werden sterische hindering en elektrostatische afstoting dominant en beperkten ze de hybridisatie van DNA-moleculen.
Oprichting van V. cholerae DNA-biosensor-kalibratiecurve
Uit het resultaat getoond in Fig. 6a, nam de DNA-biosensorrespons proportioneel toe met de toenemende cDNA-concentratie van 1,0 × 10 −21 tot 1.0 × 10 −8 M (R 2 =0,99) met een detectielimiet van 1,0 × 10 −21 M. De detectielimiet werd berekend op basis van driemaal de standaarddeviatie van de biosensorrespons bij de responscurve die de detectielimiet benadert gedeeld door de lineaire kalibratiehelling. Het brede lineaire detectiebereik van de DNA-biosensor was te danken aan de sterk monodisperse en bolvormige PSA-latexdeeltjes in een bereik van submicron dat werd gebruikt als de dragermatrix voor DNA-immobilisatie. De acrylzuurrijke laag op het oppervlak van de latexdeeltjes bood een grote bindingsplaats voor aanhechting van DNA-capture-probes om een maximaal bedekt oppervlak te creëren door de DNA-ontvankelijke laag. Bovendien versterkte de opname van AuNP's op de PSA-gemodificeerde SPE het analytische signaal van de DNA-hybridisatierespons verder, en dit zorgde voor een hoge gevoeligheid van de DNA-biosensor (Fig. 6b).
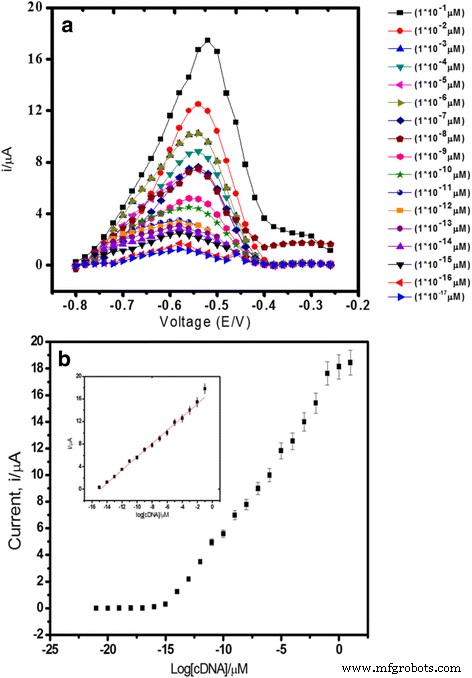
Differential pulse voltammograms (a ) and DNA biosensor linear range (b ) obtained using various cDNA concentrations from 1.0 × 10 −15 to 1.0 × 10 −1 μM V. cholerae target DNA and 5 μM signal probe
DNA Probe Immobilisation and Hybridization Times
It took about 8 h for the capture probe to be immobilised on the PSA copolymer particles surface, as illustrated by the DNA biosensor response in Fig. 7a, which showed a current increment from 1.0 to 8.0 h of capture probe immobilisation time, after which no obvious change in the DPV current was observed. Longer immobilisation time resulted in a higher amount of DNA probes immobilised onto the latex. After 8.0 h of exposure to the DNA probes, the hydrophilic functional latex with reactive carboxyl groups at the surface was presumably fully attached with the DNA probes. DNA hybridisation time, on the other hand, is the rate limiting step, which determines the response time of the DNA biosensor. Based on the DNA biosensor response trend in Fig. 7b, the response time of the V. cholerae DNA biosensor developed in this study was estimated to be about 60 min for the dual hybridisation processes to complete.
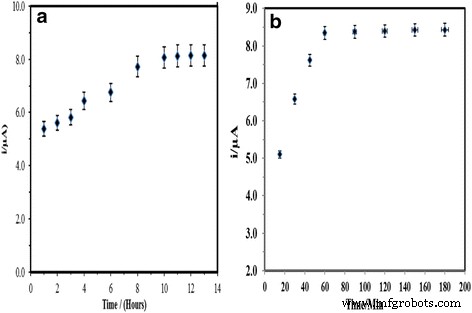
DNA probe immobilisation duration on the immobilised PSA latex colloidal particles (a ) and DNA hybridization duration of the DNA biosensor (b ) in 0.05 M potassium phosphate buffer at pH 7.0 containing 5 μM target DNA and reporter probe and 1 mM AQMS at 2.0 M ionic strength
Long-Term Stability and Regeneration of V.cholerae DNA Biosensor
Figure 8 shows the shelf life of the V. cholerae DNA biosensor. The DNA biosensor showed the highest response to the detection of 5 μM of V. cholerae cDNA for the first month of the experimental period. The electrochemical DNA biosensor was able to retain 95% of its initial DPV current after 58 days of storage period. The DNA hybridisation response was then gradually decreased to about 75% of its original response on the 75th day and exhibited 40% of its initial performance on the 100th operational day. The bioactivity of the immobilised capture probe was finally declined to 30% after 3 months of storage period. The reproducibility of each calibration point, which was repeated on five replicate DNA electrodes, gave satisfactory relative standard deviation (RSD) between 2.4 and 4.5% (n =5).
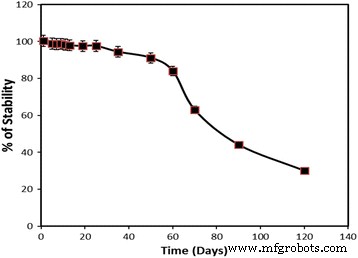
The life span profile of the fabricated V. cholerae DNA sensing electrode. The electrode was stored in 0.05 M potassium phosphate buffer (pH 7.0) at 4 °C after every DPV measurement was taken
Regeneration of biosensor indicates whether the biosensor is reusable for a series of consecutive analyses. The regeneration method used in this study was conducted based on previously reported protocol in other studies [11, 21] with slight modifications. In this study, 0.1 M of NaOH solution was used as the regeneration solution to break the hydrogen bonds between base pairs of hybridised dsDNA. With the result from Fig. 9, it is notable that the DNA biosensor response declined significantly after incubation in 0.1 M of NaOH and the percentage of the DNA biosensor response reduced from 35.1 to 5.2% relative to the DNA biosensor initial response after incubation in 0.1 M of NaOH solution from 30 to 240 s. The DNA biosensor response decreased with the increasing incubation time signifies the hydrogen bonds between hybridised dsDNA were broken up by the alkaline regeneration solution. However, rehybridisation of the DNA biosensor was able to attain almost 100% of its initial response for a consecutive six DNA analyses with a reversibility RSD of 5%.
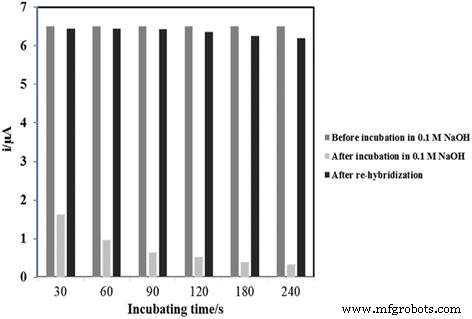
Repeatability of V. cholerae DNA biosensor using 0.1 M NaOH regeneration solution and rehybridization solution containing 5 μM cDNA and detection probe and 1 mM AQMS at 2.0 M ionic strength in 0.05 M potassium phosphate buffer (pH 7.0)
Determination of V. cholerae Bacteria with the Developed DNA Biosensor
The optimised DNA biosensor has been applied to quantify the V. cholerae DNA extracted from various V. cholerae bacterial strains. Table 3 presents the results acquired from DNA tests carried out with the hybridisation medium spiked with different strains of V. cholerae DNAs and other bacterial species at a concentration within the calibration range of the DNA biosensor. The DNA biosensor showed superior selectivity towards V. cholerae J3324–I, V. cholerae J3324–II, and V. cholerae UVC1324 with high DPV current response and low current signals were obtained for the evaluation of both Citrobacter freundii (CF-I) and Citrobacter freundii (CF-II).
Recovery of V. cholerae J3324 and V. cholerae UVC1324 DNAs at three different concentrations spiked into the hybridisation buffer demonstrated 91.4 ± 2.2% to 108.9 ± 4.8% (n =3) of recoveries percentage (Table 4). This result suggests that the proposed PSA-AuNPs-based electrochemical DNA biosensor could be adopted for highly reliable and accurate detection of V. cholerae DNA in environmental and clinical samples.
Performance Comparison with Other Reported V. cholerae DNA Biosensors
Based on the data summarised in Table 5, the proposed electrochemical DNA biosensor based on PSA-AuNPs immobilisation material shows an exceptional broad linear quantification range compared to other planar two-dimensional electrodes as the DNA supporters. This clearly demonstrates the advantage of the micro-sized latex particles where the polymeric PSA is capable to intensify the probe binding capacity with a simple loading method via the classical EDC/NHS coupling compared to avidin-biotin technology [24, 26] and ultra-low detection limit in zeptomolar range with reasonable assay time.
Conclusions
This study reports the development of an electrochemical DNA biosensor for the detection of one of the most devastating high-risk V. cholerae pathogens. The PSA-AuNPs-modified DNA biosensor can be used for direct detection of DNA of interest from the extracted DNA without the need of amplification reaction via conventional PCR method, which is commonly used in those previously reported V. cholerae DNA biosensors. In addition, no further dilution of the extracted DNA is needed as the high-capacity AuNPs-doped latex microspheres-based DNA biosensor is highly sensitive for the quantitation of DNA at extremely low level in sub zeptomolar range. Therefore, the electrochemical DNA biosensor is greatly suitable as a surveillance and diagnostic tool to control the epidemic of the fatal intestinal infection.
Nanomaterialen
- Het potentieel voor het integreren van visuele data met het IoT
- Demonstratie van een flexibele op grafeen gebaseerde biosensor voor gevoelige en snelle detectie van eierstokkankercellen
- De verbeterde foto-elektrochemische detectie van urinezuur op Au Nanodeeltjes gemodificeerde glasachtige koolstofelektrode
- Een zeer gevoelige elektrochemische DNA-biosensor van acryl-goud nanocomposiet voor de bepaling van het geslacht van Arowana-vissen
- Monodispergerende koolstofnanosferen met hiërarchische poreuze structuur als elektrodemateriaal voor supercondensator
- Verbeterde stabiliteit van gouden magnetische nanodeeltjes met poly(4-styreensulfonzuur-co-maleïnezuur):op maat gemaakte optische eigenschappen voor eiwitdetectie
- Gecontroleerde synthese van BaYF5:Er3+, Yb3+ met verschillende morfologie voor de verbetering van upconversie-luminescentie
- Een op grafeenoxide gebaseerde fluorescerende aptasensor voor de inschakeldetectie van CCRF-CEM
- Elektrisch geleidende nanovezelcomposiet van TPU met hoge rekbaarheid voor flexibele reksensor
- De fabricage van grote, uniforme grafeen-nanomeshes voor snelle, kamertemperatuur directe Terahertz-detectie
- Impossible Objects werkt samen met BASF voor composiet 3D-printen



