Decodering van de SARS-CoV-2-genomen – Oorsprong
De aanhoudende COVID-19-pandemie vormt een wereldwijde bedreiging voor de volksgezondheidsstelsels en heeft de wereldeconomie hard getroffen. De boosdoener van deze pandemie, SARS-CoV-2, is niet de typische griep. Dit virus tast zowel de bovenste als de onderste luchtwegen aan, verstoort het kernproces van het leven, de ademhaling en is daarom dodelijk. Op 6 april 2020 heeft Worldometer 1.337.166 gevallen gemeld met 74.176 doden over de hele wereld.
Het onderzoeken van SARS-CoV-2 op genoomniveau zal inzicht verschaffen in het begrijpen van de oorsprong van dit virus. Het zal wetenschappers ook helpen diagnostische instrumenten te ontwerpen om deze onzichtbare ziekteverwekker te detecteren en de uitvinding van therapieën te vergemakkelijken om het verlies aan mensenlevens tot een minimum te beperken.
Het SARS-CoV-2-genoom begrijpen
Een virus is een infectieus agens dat een levende gastheer nodig heeft om te gedijen en te repliceren. SARS-COV-2 is ook een enkelstrengs RNA-virus met een genoom van bijna 30 kb nucleotidebasen met 12 vermeende open leesramen. Kort nadat de epidemie in december 2019 begon, hebben Chinese wetenschappers het SARS-CoV-2-genoom gesequenced. Verschillende wetenschappelijke groepen hebben de afgelopen weken volledige genomische sequenties van SARS-CoV-2 vrijgegeven. Deze zijn openbaar beschikbaar in Genbank en de Coronavirus Database.
Oorsprong van het SARS-CoV-2-virus
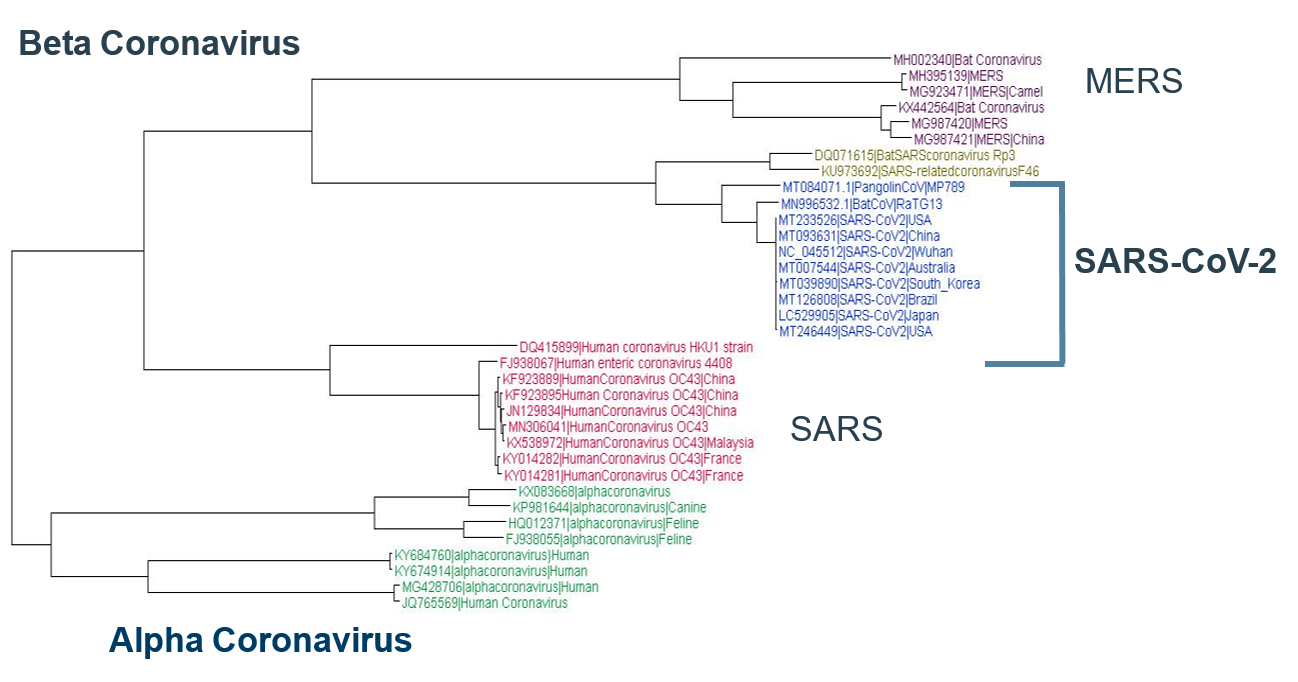
Tijdens dergelijke uitbraken kunnen niet-wetenschappelijke complottheorieën leiden tot onnodige vooroordelen tegen landen, gemeenschappen en culturen. SARS-CoV-2 is geen uitzondering, en de situatie wordt alleen maar verergerd door de razendsnelle sociale-mediaplatforms van vandaag. Het is onze plicht om deze onzichtbare vijand door een rationele wetenschappelijke lens te bekijken. Op basis van genoomanalyses is SARS-CoV-2 een virus dat van nature is geëvolueerd en is geen synthetische laboratoriumstam 1,2 . Wetenschappers hebben de volledige genomen van meer dan 100 stammen van SARS-CoV-2 verzameld uit verschillende delen van de wereld in kaart gebracht. Het blijkt dat deze stammen op nucleotideniveau voor meer dan 99,5% identiek zijn. Dit geeft aan dat de stammen niet veel muteerden in verschillende regio's, ogenschijnlijk omdat het virus al een hoge infectiegraad en virulentie heeft.
In het recente verleden hebben twee andere coronavirussen wereldwijde aandacht gekregen. Dit waren de SARS-CoV, China, 2002, en de MERS-CoV, Saoedi-Arabië, 2012. Van beide eerdere virussen werd aangetoond dat ze afkomstig waren van vleermuizen. Op basis van deze historische kennis hebben wetenschappers het coronavirus van de vleermuizen in kaart gebracht en aangetoond dat Bat CoV (RaTG13 ) was voor 96,2% identiek aan SARS-COV-2, wat de zoönotische oorsprong van laatstgenoemde bevestigt. 2 Het coronavirus gebruikt vaak een tussendrager voordat het mensen besmet. Interessant is dat rond oktober 2019 berichten over dode Maleise schubdieren met longen en longfibrosesymptomen in het Guangdong Wildlife Rescue Center in China wetenschappers ertoe aanzetten hun metagenoom te isoleren. Inderdaad, de metagenoomgegevens van de dode schubdieren bevatten het coronavirus! 3
Interessant is dat SARS-CoV-2 op het gehele genoomniveau bijna 91% identiek is aan Malayan Pangolin CoV, wat aangeeft dat Pangolins een tussengastheer zou kunnen zijn.
Wat zijn Pangolins? Het zijn mierenetende zoogdieren waar in Azië veel vraag naar is voor gebruik in de traditionele Chinese geneeskunde en voor hun vlees, dat door velen als een delicatesse wordt beschouwd. Ze zijn ook het meest verhandelde zoogdier van vandaag in de illegale handel in wilde dieren.
SARS-CoV-2 verschilt van andere bekende coronavirussen, met een sequentie-identiteit van 88% of minder. Op basis van fylogenetische analyses is SARS-CoV-2 waargenomen bij mensen, vleermuizen (RaTG13) en Maleise schubdieren een nieuwe klasse van bètacoronavirus. Bijna 35 verschillende soorten coronavirusstammen uit verschillende delen van de wereld en van verschillende organismen zijn op het hele genoomniveau geanalyseerd. SARS-CoV-2, hieronder in blauw weergegeven, is een nieuwe klasse van bètacoronavirus (Figuur 1).
Hoe komt het coronavirus de gastheer binnen?
Een van de eiwitten in het coronavirus, een Spike-eiwit genaamd, lijkt een belangrijke rol te spelen in dit proces. Het Spike-eiwit is een multifunctionele moleculaire machine die bestaat uit twee belangrijke subeenheden, S1 en S2. Het Spike-eiwit bindt eerst aan een receptor op het gastheerceloppervlak via zijn S1-subeenheid en fuseert vervolgens virale en gastheermembranen via zijn S2-subeenheid. Het domein in S1 van verschillende coronavirussen herkent een verscheidenheid aan gastheerreceptoren, wat leidt tot virale hechting. Het Receptor Binding Domain (RBD), dat uit 193 aminozuren bestaat, bindt en verbindt zich met de gastheercel. De receptor voor SARS-CoV-2 bij mensen is het Angiotensin Converting Enzyme 2 (ACE2). ACE2 zit vast aan het buitenoppervlak van celmembranen in de longen, slagaders, hart, nieren en darmen. ACE2 verlaagt de bloeddruk door de splitsing van angiotensine II, een vasoconstrictor-peptide in angiotensine1-7, een vasodilatator, te katalyseren. Helaas lijkt ACE2 ook een populair toegangspunt te zijn voor coronavirussen.
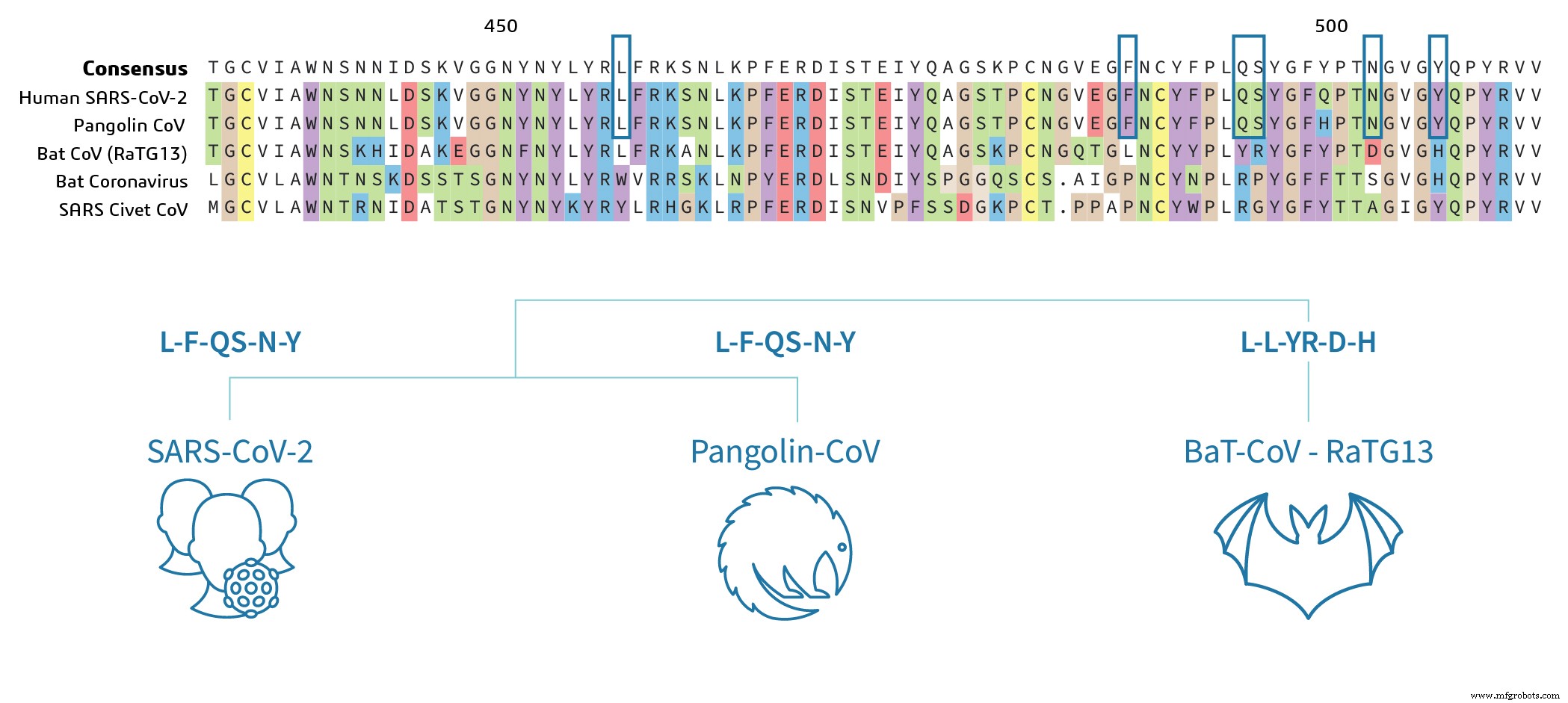
De Pangolin CoV- en SARS-CoV-2-sequenties zijn sterk geconserveerd in het RBD-gebied, wat aangeeft dat het pathogene potentieel van het virus erg vergelijkbaar is tussen Pangolin CoV en SARS-CoV-2. De belangrijkste aminozuurresiduen, die de binding bepalen, zijn identiek tussen Pangolin CoV en SARS-CoV-2 in de sequentie-uitlijning (gemarkeerd met blauwe vakjes in figuur 2a) en de belangrijkste aminozuren (LFQSNY) weergegeven boven de tekenfilms in figuur 2b . Interessant is dat de vleermuis SARS-CoV-2 RBD verschilt in 17 aminozuurresiduen, waaronder vijf kritische residuen voor binding 3 . Op basis van analyse van de sequentiegegevens kan men speculeren dat de vleermuis-SARS-CoV-2 mogelijk niet de belangrijkste residuen heeft om aan het ACE2-eiwit van de gastheercel te binden om infectie te veroorzaken. Dit vereist experimenten om te bevestigen.
Zoals eerder opgemerkt, bevat het Spike-eiwit twee functionele domeinen:een receptorbindend domein en een tweede domein dat sequenties bevat die de fusie van de virale en celmembranen bemiddelen. Het Spike-glycoproteïne moet worden gesplitst door celproteasen om blootstelling van de fusiesequenties mogelijk te maken en is daarom nodig voor celinvoer. Vergelijking van de S1/S2-splitsingsplaatssequentie van Pangolin CoV en vleermuis-SARS-CoV-2 toont een insertie van het furineherkenningsmotief. Dit duidt op een duidelijk mechanisme voor het binnendringen van het virale genoom in het cytoplasma van de gastheer voor replicatie, zoals weergegeven in figuur 3.
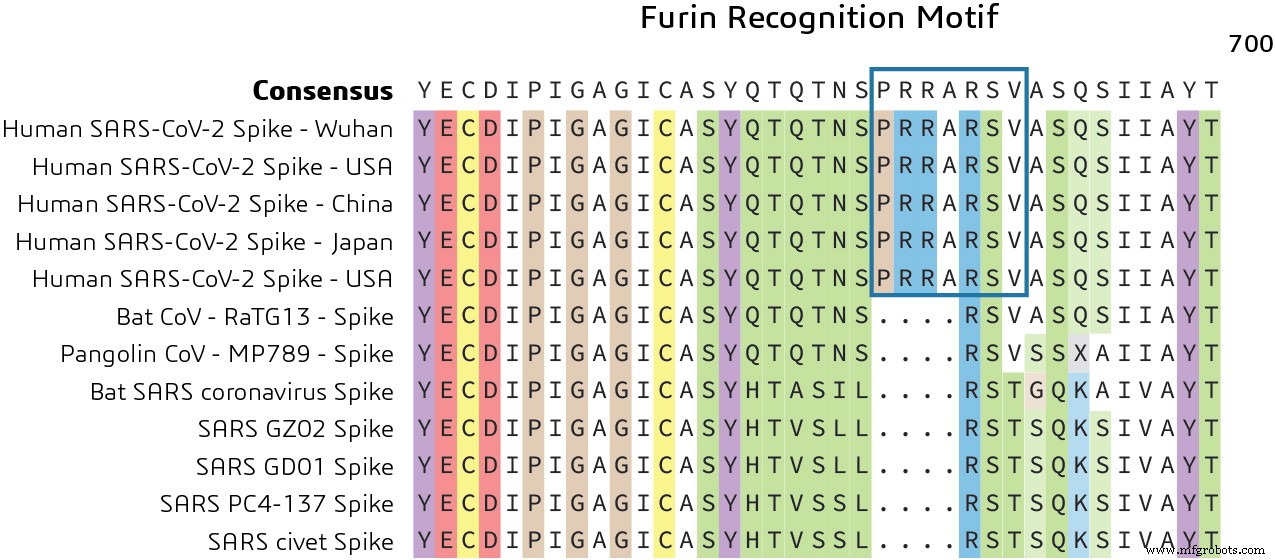
Wat is de rol van het furineherkenningsmotief? Bij mensen wordt het furine-herkenningsmotief (PRRARSV) herkend door het FURIN-eiwit, een lid van de S8-familie van subtilisine-achtige peptidasen die helpt om delen van het eiwit te verwijderen om hun conformatie te veranderen van een inactieve naar een actieve toestand.
Er is gesuggereerd dat de verwerving van deze furine-splitsingsplaats een 'functiewinst' zou kunnen zijn die een vleermuis-CoV in staat stelde om in mensen te springen en zijn huidige epidemische verspreiding te beginnen. Dit kan een mogelijke manier zijn om nieuwe medicijnen te onderzoeken die gericht zijn op het blokkeren van dit motief om de replicatie van het virus in de gastheer te voorkomen.
Zorgvuldig onderzoek van het Spike-eiwit in SARS-CoV-2 toont dus het geoptimaliseerde RBD, een furineherkenningsmotief, zoals sommige MERS-coronavirussen, en het vermogen ervan om sterk aan het ACE2-eiwit te binden. Dit suggereert dat er een natuurlijk selectieproces in het spel is. Het is aangetoond dat natuurlijke recombinatiegebeurtenissen in virussen die een gastheer co-infecteren, hun gastheerbereik verbeteren, terwijl ze ook de virulentie en virusadaptatie verhogen. SARS-CoV-2-genoomgegevens met ruggengraat van de vleermuis (RaTG13) en pangolin CoV geven opnieuw aan dat dit een virus is dat wordt gegenereerd door natuurlijke recombinatie .
Wat is de directe donor van SARS-CoV-2 aan mensen?
De SARS-CoV-2-sequentie heeft een mix van zowel vleermuis-SARS-CoV (RaTG13) als gebieden van geconserveerde Pangolin CoV die alleen kunnen plaatsvinden tijdens recombinatie van deze virale genomen. Ook houdt een functiewinst, zoals te zien is bij het furineherkenningsmotief, een andere virusrecombinatie in. Om recombinatie te laten plaatsvinden, is het niet meer dan logisch dat er een natuurlijke gastheer is die deze virale genomen herbergt. Is het een ander schubdier? Of een ander wild dier op de Wuhan-zeevoedselmarkt? Dit is nog niet bekend. Inzicht in de oorsprong kan toekomstige uitbraken van virusstammen en wereldwijde pandemieën helpen voorkomen.
Neem voor meer informatie contact met ons op:https://www.3dsbiovia.com/about/contact/.
Biologie
- De 555 IC
- De kwadratische formule
- Decodering 'Industry 4.0'
- Een supply-chain-aanpak om de coronavirus-uitdaging op te lossen
- Zal de coronavirusepidemie dienen als een wake-up call voor wereldwijde toeleveringsketens?
- Het coronavirus vernietigt traditionele toeleveringsketens
- Zes supply chain-strategieën voor olie en gas in het coronavirustijdperk
- Het coronavirus kan een einde maken aan slechte verzendgegevens
- Hoe datawetenschap de uitbraak van het coronavirus heeft helpen bestrijden
- 5 Ws van de SARS-CoV-2 RapidPlex-sensor
- De oorsprong van de Crippa Tube Bender



